Actinobacteria
#Archaea
#Archaea
활성화 함수




2차 면역반응
#Archaea
 1. snRNPs 5개 혹은 ribonucleoproteins 접근
2. intron에 붙음
3. intron의 양 끝(5', 4')이 모이면서 고리를 생성
4. intron이 제거됨
- intron은 이후 다른 과정에 참여함
6. snRNPs는 다시 재활용됨
1. snRNPs 5개 혹은 ribonucleoproteins 접근
2. intron에 붙음
3. intron의 양 끝(5', 4')이 모이면서 고리를 생성
4. intron이 제거됨
- intron은 이후 다른 과정에 참여함
6. snRNPs는 다시 재활용됨
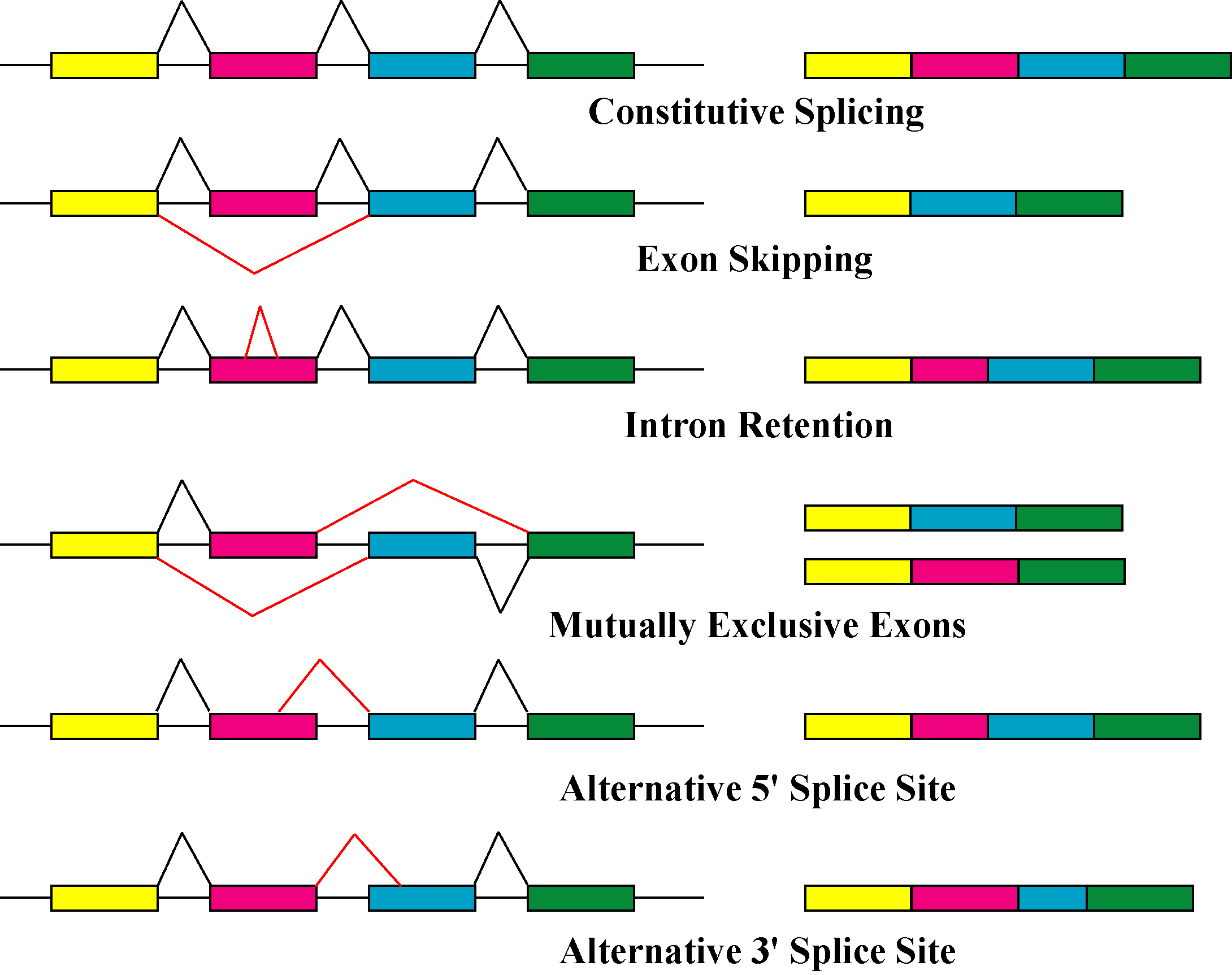 ## Exon Skipping (Cassette Exon)
- 특정 exon을 건너 뛰어 버린다.
## Intron Retention (Retained Intron)
- intron이 중간에 그대로 끼어있음
## Mutually Exclusive Exon
- 특정한 exon에서 여러 돌연변이가 발생해 다양한 종류의 돌연변이를 야기한다.
## Alternative 5' Donor Site
- upstream exon의 중간부분이 연결된다.
## Alternative 3' Acceptor Sites
- downsteram exon의 중간부분이 연결된다.
## Exon Skipping (Cassette Exon)
- 특정 exon을 건너 뛰어 버린다.
## Intron Retention (Retained Intron)
- intron이 중간에 그대로 끼어있음
## Mutually Exclusive Exon
- 특정한 exon에서 여러 돌연변이가 발생해 다양한 종류의 돌연변이를 야기한다.
## Alternative 5' Donor Site
- upstream exon의 중간부분이 연결된다.
## Alternative 3' Acceptor Sites
- downsteram exon의 중간부분이 연결된다.
#Allele_Drop-Out
antisense RNA
Area Under the Receiver Operator Curve
B cell receptor
β-selection
T cell의 전구세포는 조혈모세포-척수-흉선을 따라 이동하게 된다.
대부분은 αβ T세포가 되지만 5% 정도는 γδ TCR을 가진 세포들이 된다.
흉선의 피질과 수질로 구분되어 있으며, T cell의 전구세포(흉선세포) 성장은 흉선의 기질세포들과 상호작용하며 일어난다.
초기의 흉선세포들은 CD4, CD8 어느 단백질도 띄지 않고 있기 때문에 double negative(DN)세포라고 부른다.
흉선세포의 DN기는 크게 4 단계로 나뉘어지며, 이때는 CD44, CD25 같은 단백질들을 띄게 된다.
DN3기에서 DN4기로 넘어갈 때, V(D)J 유전자 재조합의 결과 β-chain을 형성할 수 있는 T cell 만이 선택되는 β-selection이 일어난다.
선택받은 T cell 만이 다음 단계를 넘어서 성장하게 된다.
canonical correlation analysis
세포지도
Single cell 연구의 장점은 각 세포들의 '상태'를 하나하나 알 수 있다는 것이며, 이러한 '상태'들을 cell state라고 부른다.
Time series experiments of differentiation have observed cells transitioning between a starting state and one or more end states, with many cells distributed along a “trajectory” between them.
세포의 발달과정, 상태변화과정을 말한다.

염색질 접근성
#Soft_Clipping #Hard_Clipping
Convolutional Neural Network
 - 연산으로는 다음과 같이 나타낼 수 있다.
$$\huge f(x) * g(x) = h(x)$$
*f = filter, g= feature map, h= output*
# Deconvolution
- Deconvolution은 Convolution의 반대과정이다.
- 연산으로는 다음과 같이 나타낼 수 있다.
$$\huge f(x) * g(x) = h(x)$$
*f = filter, g= feature map, h= output*
# Deconvolution
- Deconvolution은 Convolution의 반대과정이다.
https://dambaekday.tistory.com/3
#Convolution #Deconvolution
Copy Number Variation
https://github.com/google/deepvariant
"DeepVariant is a deep learning-based variant caller that takes aligned reads (in BAM or CRAM format), produces pileup image tensors from them, classifies each tensor using a convolutional neural network, and finally reports the results in a standard VCF or gVCF file."
Depth First Search

#Algorithm
#tool
highly variable genes
Intrinsically Disordered Protein
면역 레퍼토리
"Immune repertoire refers to all of the unique T-cell receptor (TCR) and B-cell receptor (BCR) genetic rearrangements within the adaptive immune system."
Basic Training
YouTube Playlist(7hrs): Community Nextflow & nf-core Foundational Training
nextflow info
process takes input data and give output data
process includes command or script which has to be executedchannel is where the data come in#!/usr/bin/env nextflow
params.greeting = 'Hello world!'
greeting_ch = Channel.of(params.greeting)
process SPLITLETTERS {
input:
val x
output:
path 'chunk_*'
script:
"""
printf '$x' | split -b 6 - chunk_
"""
}
process CONVERTTOUPPER {
input:
path y
output:
stdout
script:
"""
cat $y | tr '[a-z]' '[A-Z]'
"""
}
workflow {
letters_ch = SPLITLETTERS(greeting_ch)
results_ch = CONVERTTOUPPER(letters_ch.flatten())
results_ch.view { it }
}
nextflow run hello.nf
#N E X T F L O W ~ version 23.10.0
#Launching `hello.nf` [confident_mclean] DSL2 - revision: 197a0e289a
#executor > local (3)
#[bd/de1214] process > SPLITLETTERS (1) [100%] 1 of 1 ✔
#[cc/562f4f] process > CONVERTTOUPPER (1) [100%] 2 of 2 ✔
#WORLD!
#HELLO
Also known as third-generation sequencing
Long Short-Term Memory
micro RNA
Mutual Nearest Neighbors
mRNA를 만드는 과정: Capping → Tailing → Splicing → RNAediting
이형상실/이형접합성 소실

Reference allele는 G, alternative allele는 A가 확인된 샘플.
https://www.genome.gov/genetics-glossary/Frameshift-Mutation
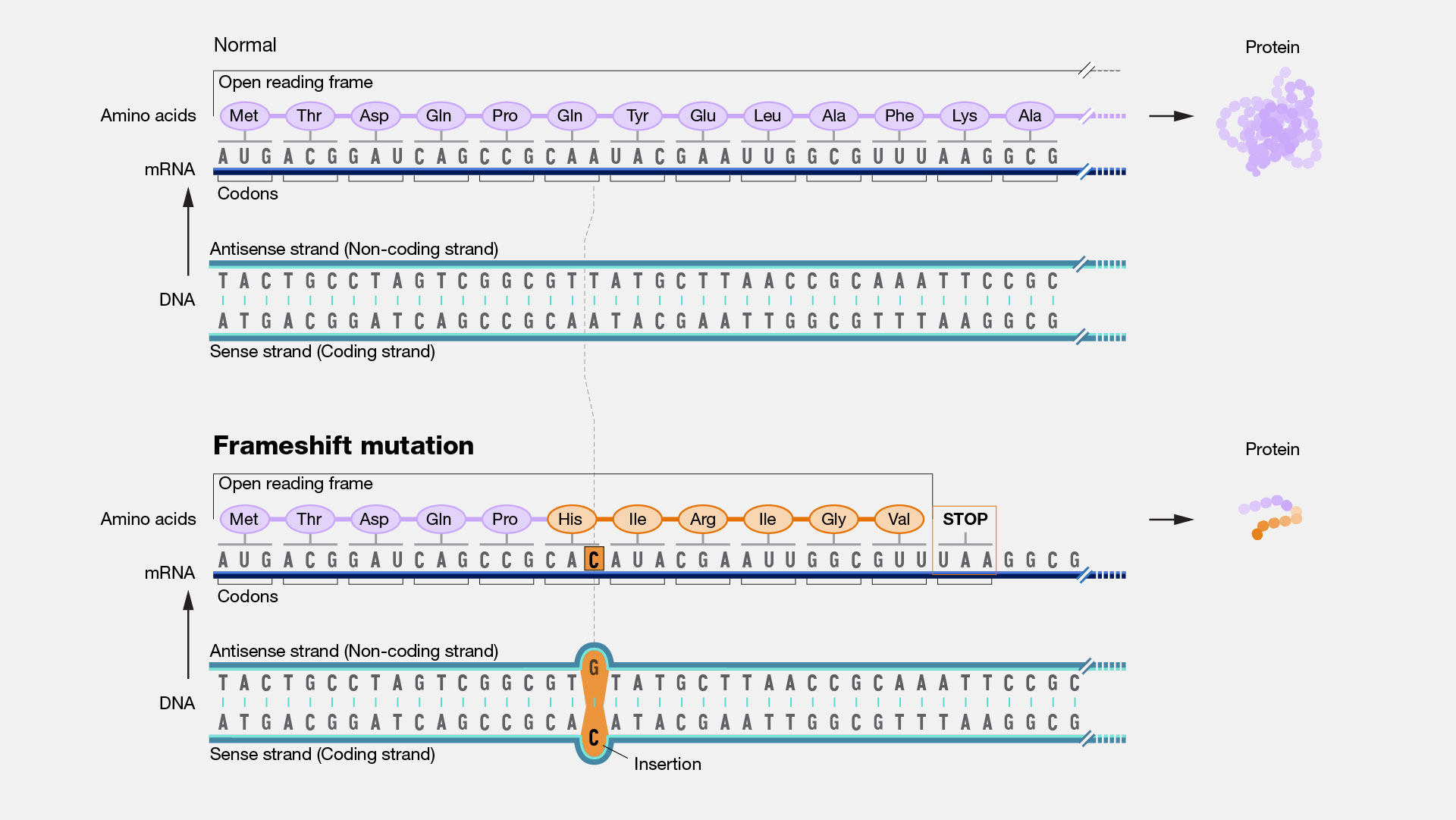
Next Generation Sequencing
Non SQL / Not Only SQL
Non-Relational DataBase
Open Reading Frame


Operational Taxonomical Units
Pearson's chi-squared test
piwi interacting RNA
RNA binding proteins
Relational DataBase, 관계형 데이터베이스
CREATE TABLE customers(
id INT NOT NULL ATUO_INCREMENT,
name VARCHAR(255) NOT NULL,
email VARCHAR(255) NOT NULL,
phone_number VARCHAR(255) NOT NULL,
PRIMARY KEY (id)
);
CREATE TABLE orders(
id INT NOT NULL AUTO_INCREMENT,
customer_id INT NOT NULL,
product_id INT NOT NULL,
quantuty INT NOT NULL,
price INT NOT NULL,
PRIMARY KEY (id),
FOREIGN KEY (customer_id) REFERNCES customers (id),
FOREIGN KEY (product_id) REFERENCES products (id)
);
orders.id,
orders.customer_id,
orders.product_id,
orders.quantity,
orders.price,
products.name
From oders
INNER JOIN products
ON orders.product_id=products.id
WHERE orders.customer_id=1;
Row Echelon Form matrix, 사다리꼴 행렬
Rectified Line
mRNA
rRNA
tRNA
snRNA
snoRNA
aRNA
miRNA
siRNA
piRNA
ribosomal RNA
Single Cell RNA Sequencing
Shapley Additive Explanations
small interfering RNA
Single-molecule Fluorescene in situ Hybridization.
Single molecule real time sequencing from PacBio
small nucleaolar RNA
small nuclear RNA
small nuclear ribonucleoproteins
SELECT "Hello, World!";
T cell receptor
흉선.
DFS는 깊이우선탐색으로, tree structure의 가장 바닥이 어디인지 빠르게 파악하는데 도움을 준다.
DFS를 활용한 traversal의 경우, 어디를 방문 시작점으로 잡느냐에 따라 결과물이 달라진다.
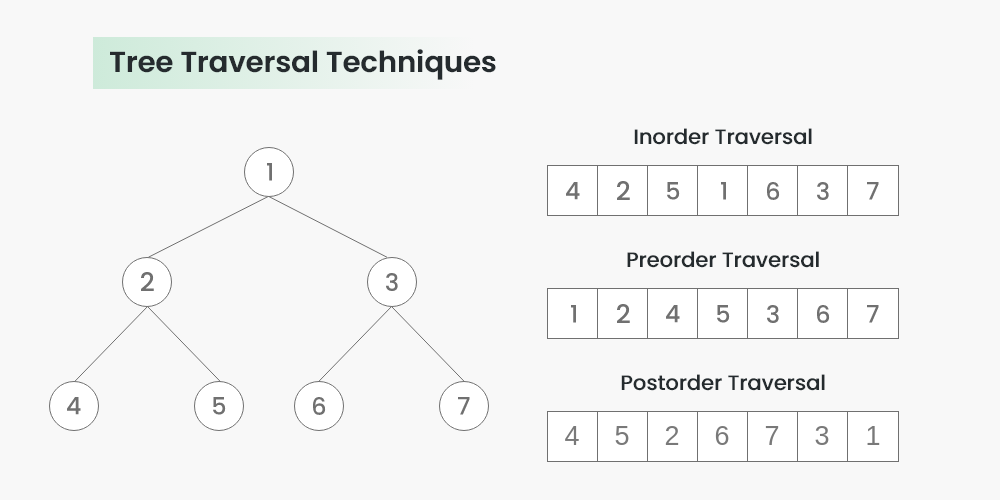
다시금 말하지만 DFS에서 중요한 것은 '가장 바닥이 어디인지', '이 tree구조의 깊이가 얼마나 되는지' 빨리 파악하는 것에 초점을 둔다.
위의 그림에서 가장 왼쪽 끝 바닥의 node인 4의 위치가 항상 앞쪽에 나열된다는 점에 주목하자.
# Binary Search Tree(BST) Structure
# 0. Build BST node
Class Node:
def __init__(self, key):
self.left = None # child 1
self.right = None # child 2
self.val = key
# 1. Inorder Traversal
def printInorder(root):
if root:
printInorder(root.left) # Recursive Function. Keep go to left!
print(root.val, end=" ") # Come back to root
printInorder(root.right) # Go to right
# 2. Preorder Traversal
def printPreorder(root):
if root:
print(root.val, end=" ") # Root First
printPreorder(root.left) # Recursive Function. Keep go to left!
printPreorder(root.right) # Now go to right
# 3. Postorder Traversal
def printPostorder(root):
if root:
printPostorder(root.left) # Recursive Function. Keep go to left!
printPostorder(root.right) # Go to right
print(root.val, end=" ") # Check the root as last
# Driver code
if __name__ == "__main__":
root = Node(1)
root.left = Node(2)
root.right = Node(3)
root.left.left = Node(4)
root.left.right = Node(5)
# Function call
print("Inorder traversal of binary tree is")
printInorder(root) # 42513
print("Preorder traversal of binary tree is")
printPreorder(root) # 12453
print("Postorder traversal of binary tree is")
printPostorder(root) # 45231
transfer RNA
V(D)J는 항체의 상단의 variable region을 말한다.
Light Chain: V-J
Heavy Chain: V-D-J
이렇게 V-(D)-J 영역을 구성하는 단백질을 유전자 재조합 과정을 통해 바꿔가기 때문에, 항체들은 Immune Repertoire를 넓게 구성할 수 있는 것이다.
그리고 이때 관여하는 수많은 단백질들 중 RAG1, RAG2, Ku, Artemis, DNA Ligase 같은 단백질들이 있다.
-> 따라서 V(D)J 유전자 재조합은 BCR, 혈청 내 항체, TCR 3개 모두에 영향을 끼친다.
Whole-Exome Sequencing
Zinc Finger
#file_type
#file_type
#file_type
#file_type
variant calling file
Click here to access Cosma's documentation
| Space | Re-run the force-layout algorithm |
| S | Move the cursor to Search |
| Alt + click | (on a record type) Deselect other types |
| R | Reset zoom |
| Alt + R | Reset the display |
| C | Zoom in on the selected node |
| F | Switch to Focus mode |
| Escape | Close the active record |
Version 2.0.4 • License GPL-3.0-or-later